Nowy panel MTHFR UK cz.1
wrzesień 2, 2019 by Jarek
Kategoria: MTHFR i inne polimorfizmy
Pragmatyzm ponad wszystko, takie powinno być hasło twórców paneli genetycznych. Dlaczego? Bałagan i wybór maksymalnie dużego panelu nie wniesie niczego nowego, a wiedza sumaryczna, skondensowana może być łatwiejsza do “strawienia” i zrozumienia….i w końcu MTHFR UK to zrozumiał.
Pierwsza uwaga:
Panel jest obecnie czytelnie pogrupowany na tematy najbardziej istotne populacyjnie w miarę uporządkowany sposób.
Druga:
Mamy działy, a te mają swoje specyficzne podgrupy co powoduje, że świetnie się to sczytuje.
Trzecia:
Koniec z zielonym kolorem na panelu! Mamy biały, żółty i czerwony. Oznacza to, że jedynie zmiana/odchylenie od standardu jest tutaj prezentowane i podkreślane.
Czwarta:
Kolejność i logika panelu jest obecnie inna. Mogę się domyśleć, że skoro jelita są najważniejsze i alergeny to temat jelit jest pierwszy i najważniejszy. Potem mamy konsekwencje przyjętej metodologii. Skoro jelita są pierwsze to stan zapalny jest drugi. Po nim zaś :
*detox
*mitochondria, które są z detoxem ściśle związane
*metylacja, która jest powiązana z każdym z tych tematów
*w konsekwencji neuroprzekaźniki
*na końcu dieta i składniki odżywcze
Piąta:
Wciąż nie rozumiem logiki, która stawia kwestię absorpcji witaminy B12 (TCN, FUT2) w ujęciu jako czynnik dietetyczny, gdy w kwasie foliowym ten mechanizm jest w metylacji. Ułożenie genów odpowiadających za funkcjonowanie witaminy B12, wciąż jest dla mnie nie przemyślane jak zestawimy to z tym co jest zrobione z witaminą B9.
Szósta:
Pojawiają się nowe geny, które stanowią świetne uzupełnienie tego co do tej pory było i zwrócę na to uwagę przy analizie przykładu.
Siódma:
Pojawiają się nowe allele, ale i znika część z nich. Żałuję bardzo odcięcia genów autystycznych takich jak CACNA1C z tej analizy.
Ósma i ostatnia:
WRESZCIE układ graficzny, układ strony jest na tyle odpowiedni, że da się to objąć w krótkim czasie i nie ma konieczności poszukiwania stron, gdzie ukryto dany gen. W poprzedniej wersji było tego za dużo po prostu.


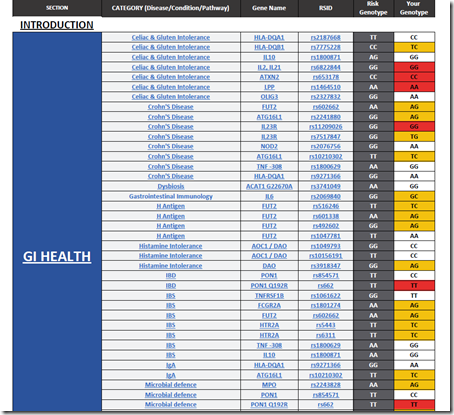
Zastanawia mnie dlaczego dla genow o tej samej nazwie sa rożne “rs” w roźnych badaniach.